Self-Supervised Graph Transformer on Large-Scale Molecular Data
Abstract
研究分子的表示学习,最近的工作直接使用GNN来进行。然而有两点问题:1.分子标注量不够2.对新合成分子泛化性不够。这篇工作提出了GROVER,graph representation from self-supervised message passing transformer. GROVER设计了node/edge/graph层级的自监督任务,并将message passing network与transformer融合,获得更好的分子编码。GROVER含有一亿参数在千万分子上进行无监督训练,在11个benchmark上达到了平均6%的精度提升。
Introduction
GNNs在分子数据上有很好表现,比如分子性质预测、虚拟筛选。虽然但是,还有摘要中提到的两点缺陷。因为分子的标注获取是比图像困难很多的,现有benchmark的标注都是远远不够的。NLP中自监督学习可以带来很大泛化能力的提升,SMILE将分子转化为序列化表示,并使用Bert风格的模型来进行分子表示预训练,N-gram graph: Simple unsupervised representation for graphs, with applications to molecules.用Ngram的方法得到边表示。但这些方法没有关注拓扑结构。
也有构建三个自监督任务来做分子预训练的,contex prediction,node masking,graph property prediction. 这些任务并不是最优的,mask任务中它们将atom种类作为label,但比起NLP的词库,atom的种类太少,频率高的atom将会极大影响编码。在graph层级的任务则是有监督的。这对下游任务造成了负迁移的可能。
GROVER设计了两种自监督任务,node层级GROVER随机掩码目标node/edge的局部子图然后用graph嵌入预测语义性质,graph层级GROVER根据领域知识抽取语义样式,然后根据graph嵌入预测这些样式是否出现。
在下游任务中,GROVER在分类任务上相对N-gram替身了22.4%,相对之前的自监督框架提升了7.4%。进一步在11个常见的benchmark上GROVER提升了平均6%.
Related Work
Self-supervised Learning on Graphs
最近在分子数据集上的是N-gram和Pre-training graph neural networks。N-gram用短距离游走以及节点嵌入来获取图表示。Pre-training则分离了node/edge上高相关的context预测任务,造成难以保持局部结构和节点特征的领域信息。(这说的什么寄吧,反正就是效果不好呗)其次它graph层级的任务是有监督,这已经说过了。
Molecular Representation learning
新的工作基本都是基于GNN的。
Transformer和GNN的背景知识
这个不懂的自己查阅,GNN相比NN会用到一个邻接矩阵,Transformer是做sequence to sequence的,整体是一个encoder-decoder的结构,encoder对序列进行编码,decoder从编码中不断生成新的预测,形成预测序列。这个过程还用到自回归的机制,后面的预测依赖于先前的预测。

message passing neural networks包含了两个参数层数L和hop数\(K_l,l=1,\cdots,L\),L决定了readout中的表示数量,hop数决定了每个L会编码几阶邻域的信息。
The GROVER Pre-training Framework
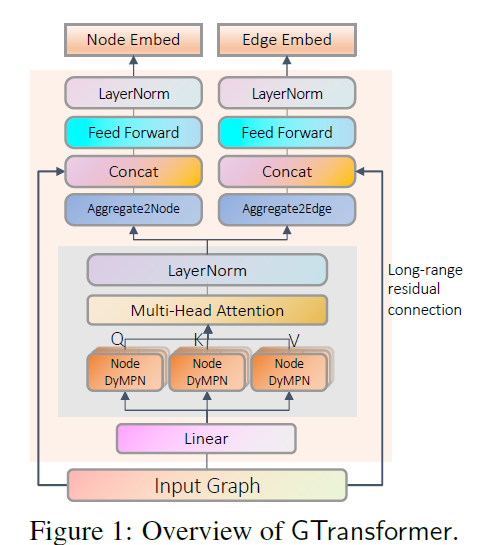
模型结构细节
GROVER包含两个模块,node/edge两块,这里只介绍node部分。重点是dyMPN模块,mesage passing有两个参数:迭代次数L和跳跃次数\(K_l,l=1,\cdots,L\)跳跃次数与图卷积的感受域有很大相关。对于给定的层数L,作者发现事先决定好的跳跃次数无法对多个数据集有效。因此,GROVER在训练时对每一层使用了随机的跳跃次数。有两种随机策略是有效的1.(a,b)间的均匀分布2.由(a,b)区间规范的正态分布。改动其实只有这里而已,其余部分都和transformer的模块一样,看Figure 1即可。
自监督任务
自监督的核心就是设计好的自监督任务,在这篇工作不使用任何有监督,以免引入不够通用的先验,而造成负迁移。这里有两种自监督任务,contextual property prediction和graph-level motif prediction,Figure2展示了这两个任务。
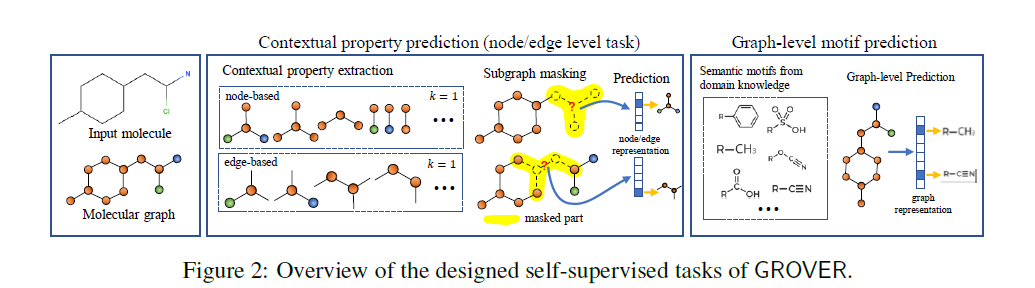
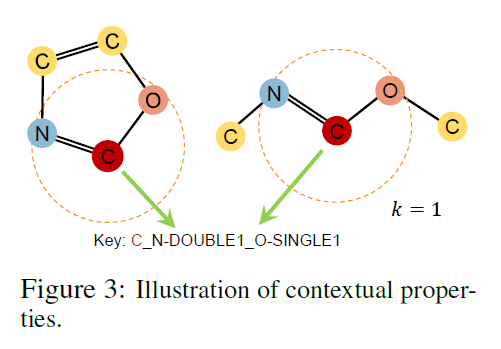
Contexual Property Prediction. 自监督任务需要满足两个条件:1.任务标签要易于获取2.任务需要反映node/edge的上下文信息。以Figure 3为例,1.给定一个目标节点(碳原子),提取它的局部子图作为k-hop邻nodes/edges,k=1时包含氮原子和氧原子,双键和单键。2.抽取子图的统计信息,具体来说,数center node的(node,edge)对,然后以字母顺序列出所有node-edge对:C_N-DOUBLE1_O-SINGLE1. 这相当于按照pair构成的Attirbute进行聚类。得到这些任务标签后,给定一个分子图,我们用GROVER抽取所有node/edge的嵌入。假定随机选择一个原子v,对应嵌入为\(h_v\)。GROVER不去预测v的种类,而是用嵌入预测v的上下文信息(即上面定义的任务标签)
Graph-level Motif Prediction. 对于分子来说,最重要的样式之一是官能团,它编码了重要的领域知识,并且可以轻易地由专业工具获取。这个任务是一个多标签分类,即判断多个样式在分子中的出现情况。
Fine-tuning for Downstream Tasks
下游任务大致可以分成三种,node/edge/graph层级。以graph层级为例,用GROVER抽取node/edge嵌入,然后用readout去得到整张图的嵌入,最后用一个额外的MLP去预测分子性质。也可以使用有监督数据去微调几轮整个encoder和readout,MLP.
实验
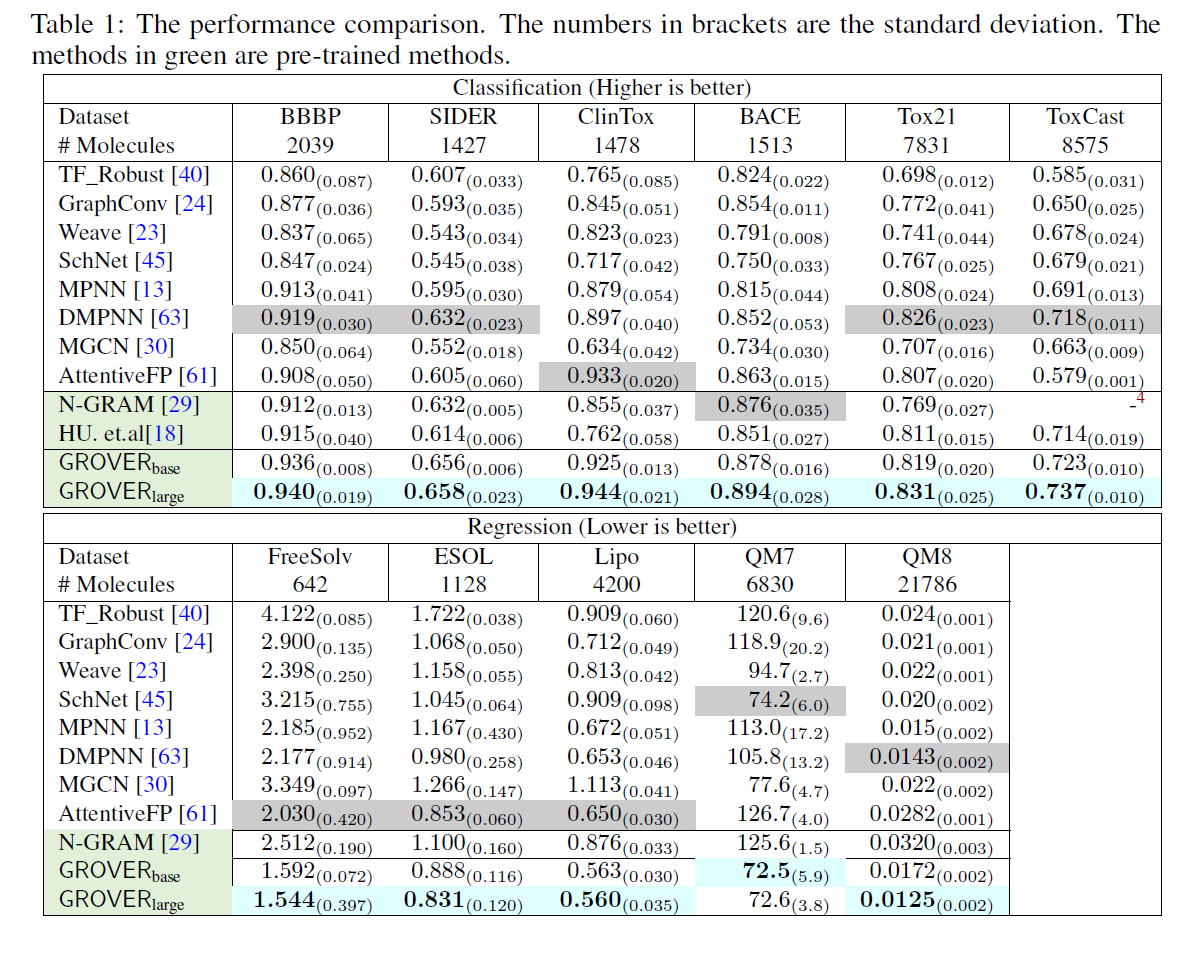
有两个GROVER模型: \(GROVER_{base},GROVER_{large}\)base有48M参数,large有100M参数,在11个任务上都效果很好。
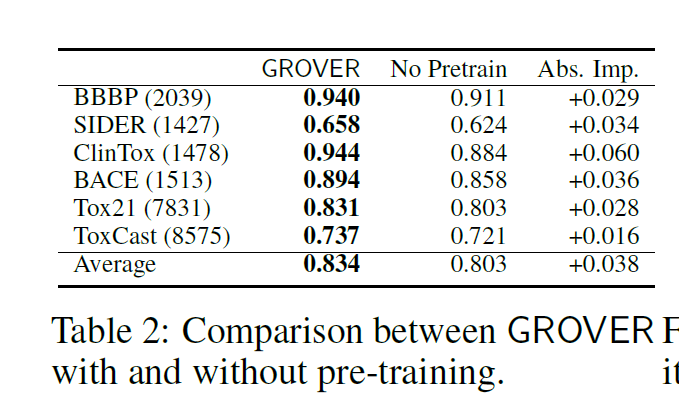
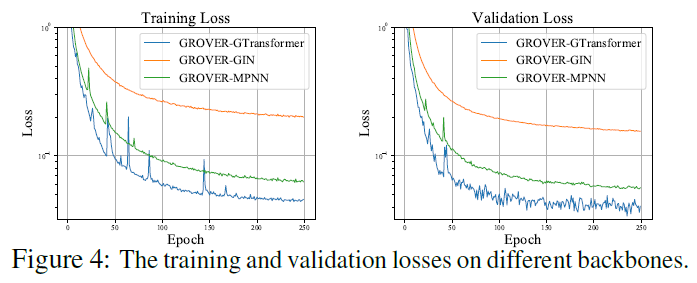
Table2对比了是否进行预训练的模型性能,在六个benchmark上进行的实验,平均分类精度会降低3.8%. Figure4对比几个backbone的性能区别,使用了一个toydataset,差不多600k未标注分子,然后保证差不多参数量(38M),GTransformer能够获得更低的训练/验证损失。
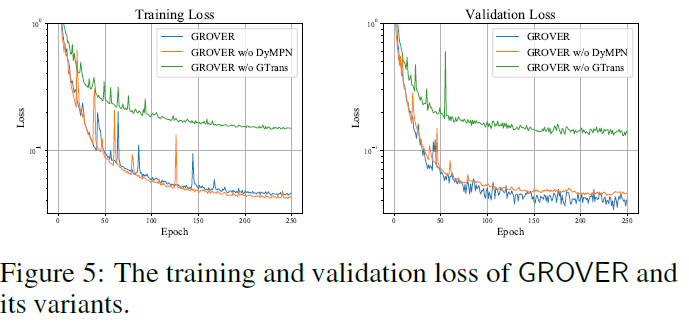
Figure5仍然是消融实验,w/o dyMPN表示用固定hop数,w/o GTrans表示用原始的Transformer。w/o GTrans的效果很差,w/o dyMPN在验证损失上稍微好一点。
总结
这篇工作主要的贡献在于设计两个自监督任务,但是实验部分没有单独讨论两种任务各自的效果。个人认为抽取官能团样式这个任务,即graph层级的自监督任务比较有用。另外一个听起来不是非常convincing。dyMPN的贡献聊胜于无,在消融实验里提升不是很大。我一直在构思新的GNN模型,我们知道CNN为邻域赋不同的权重,然后重复地在不同邻域进行aggregation。但是GNN不是这样的,每个邻域节点其权重是一样的,即使GAT可以使权重不同,它的计算代价也太大了。GAT的赋权方式是动态,而不是diversity。目前CV和NLP的模型都已经统一到Transformer的框架上,图学习相对来说还是独立的。